ITS2序列作为一种新型的DNA条形码应用于鉴定药用植物种类外文翻译资料
2023-01-10 16:01:40
ITS2序列作为一种新型的DNA条形码应用于鉴定药用植物种类
Shilin Chen1*, Hui Yao1, Jianping Han1, Chang Liu2, Jingyuan Song1*,Linchun Shi1, Yingjie Zhu1, Xinye Ma1, Ting Gao1, Xiaohui Pang1, Kun Luo3, Ying Li1, Xiwen Li1, Xiaocheng Jia1, Yulin Lin1, Christine Leon4
1中国医学科学院中国协和医科大学药学院,北京,中华人民共和国,2李嘉诚教员医学院,香港大学,香港,中华人民共和国,3药房、湖北中医药大学、武汉、湖北、中华人民共和国,4英国皇家植物园丘,里士满,英国
|
摘要 背景:植物工作组的团体为生命条形码建议两位点rbcL基因和 matK基因组合作为植物条形码,该组合被证明能从907样本中成功区分550种,在物种水平的鉴别概率约为72%。该团体认为,这两个位点的条形码是远远不够完善的。由于低识别率,探索还未结束。 方法/主要发现:在这里,我们比较了七个候选DNA条形码(psbA-trnH、rbcL、matK、rpoC1,ycf5,ITS2,及其药用植物)。我们的排名标准包括扩增效率、差分帧内和种间的差异、DNA条形码技术差距。我们的数据表明,核糖体DNA第二内部转录间隔区(ITS2)是DNA条形码技术应用最合适的区域。此外,我们使用了属于753个不同的属及4800个不同种的6600多种植物样本来测试ITS2的鉴别能力,发现在物种水平上ITS2鉴定的成功率为92.7%。 结论:ITS2区可作为一个标准的DNA条形码用于鉴定药用植物及与其密切相关的物种。我们也建议ITS2序列作为一种新的更广泛的通用条码用于识别植物类群的范围。 引文:陈、姚、韩、刘、宋、等(2010)对ITS2区验证作为一种新型的药用植物DNA条形码鉴定方法。 公共科学图书馆5(1):e8613.doi:10.1371/journal.pone.0008613 编辑:托马斯·吉尔伯特,尼尔斯玻尔研究所、生物所,丹麦 2009年9月30日收到,2009年12月8日接受,2010 年1月7日出版 版权所有:2010陈等,这是根据知识共享署名许可的条款分发的开放访问的文章,允许无限制地使用、分配和复制在任何媒介,提供原始作者和出处记。 资金来源:这项工作是由科学技术的国际合作项目(编号2007DFA30990)和特别基金支持并授予S.L.C.卫生部(编号200802043)这项工作也被来自香港研究资助局(HKU7526/06M)作为C.L.项目资助。投资者没有参与研究设计、数据收集和分析、决定发布或手稿准备。 竞争利益:作者声明,不存在竞争利益。 *电子邮件:slchen@implad.ac.cn(SC);songjingyuan@hotmail.com(JS) |
引言
据世界卫生组织估计,世界上的80%的人口在使用传统的药物来治疗疾病(http://www.worldwildlife.org/what/globalmarkets/wildlifetrade/faqs-medicinalplant.html)。有越来越多的针对药用植物的国际市场,其作用于草药和医药产品。药用植物的植物类群和密切相关的物种覆盖范围较广。
据中国的调查,药用植物共有11146种、383属、2309科,具有较丰富的生物多样性。实现在国际规模上的药用植物贸易的困难在于对这些工厂进行准确、快速的伪品认证。我们的目标是提供一种实用的,在鉴定药用植物及其伪品方面具有强大功能的工具,并确保其在贸易中能安全使用。
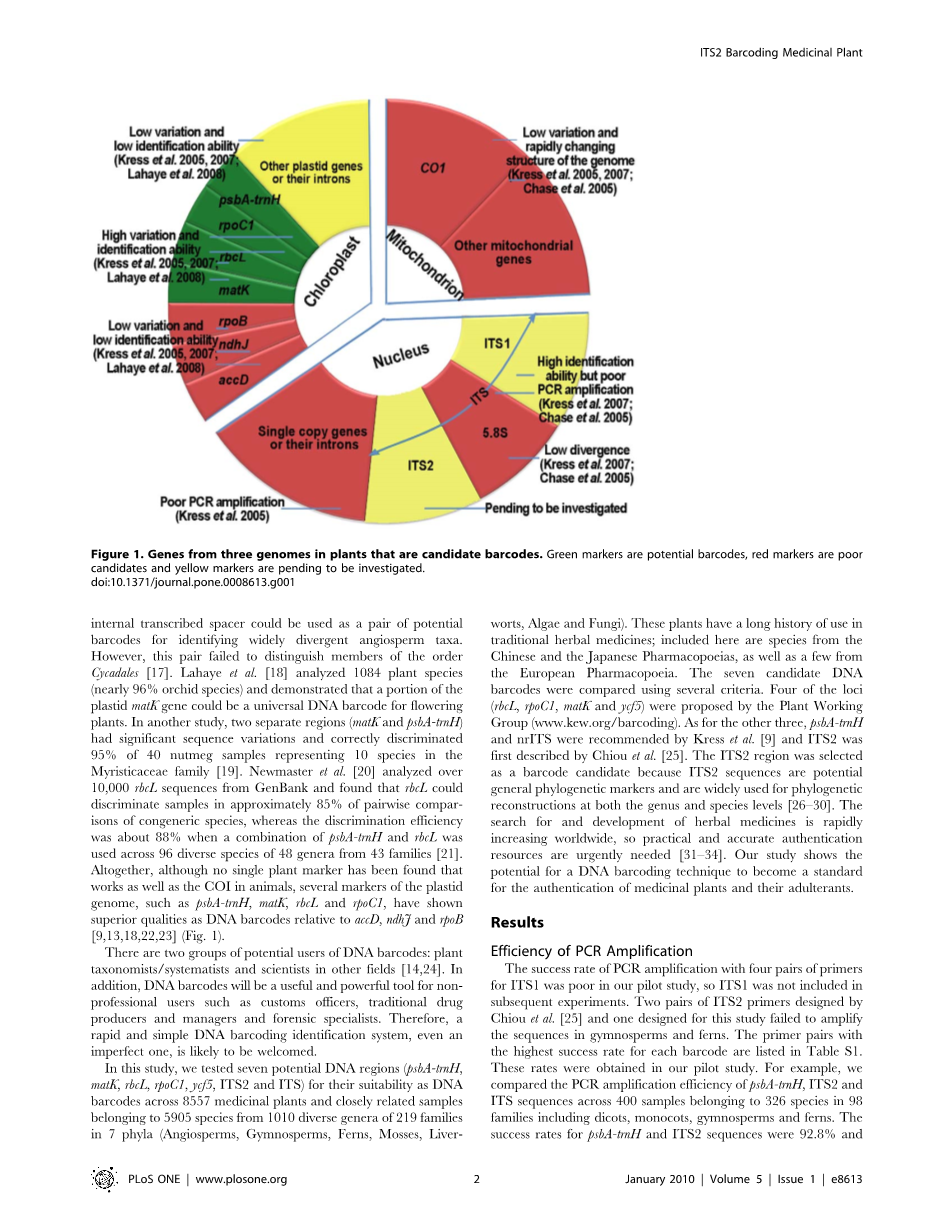
全球物种鉴定术语”DNA条形码”是首先由赫伯特在2003年[1,2]使用,并引起了全世界科学界的注意[3-7]。动物,植物和真菌的识别已使用这种技术[ 8,12 ]。大多数研究者都认为,在大多数动物物种,甚至在一些的DNA条形码真菌物种,包括那些基团子囊菌、担子菌门与壶菌门中,线粒体基因编码细胞色素C氧化酶亚基1是一个有利的片段区域。然而,在广泛类群中识别植物种类低量的基因变异,以及可变线粒体基因组[9,13-16]的结构时,CO1基因和其他植物线粒体基因的实用性是很有限的。因此,筛查适当的DNA条形码单个或多个区域,在植物细胞核和质体基因组的研究中一直是重要的研究热点(图1)。
首先,大多数的单拷贝基因在核基因组中,以及其内含子,因为候选条形码候选人缺乏通用引物对它们的扩增,都被排除在外[9]。然而,除了5.8 s,核糖体DNA的内转录间隔区(ITS)和ITS区域可能是潜在的条形码[9、14](图1)。其次,广泛的研究集中在基因和叶绿体基因组的内含子上。
例如,克勒斯等[9]通过比较10个位点来认证亲缘关系密切的7类植物科及隶属于88属53科的99种植物,他们的报告显示,psbA-trnH间隔和内转录间隔可以作为一对潜在条形码,用于识别大相径庭的被子植物类群。然而,这并未能区分成员苏铁目的秩序[17]。Lahaye等[18]分析了1084种植物(近96%的兰花品种),并证明了的一部分质体matK基因可能是开花植物的通用DNA条形码。在另一项研究中,两个独立的区域(matK序列和psbA-trnH)有显著序列变异,能正确区分肉豆蔻家庭中代表10个种的40个肉豆蔻样本,正确率为95%[19]。Newmaster等[20]分析过GenBank中的10000个rbcL基因,发现rbcL基因能对同属种的约85%的成对比较样本进行区别,而针对96种不同种类的48属43种psbA-trnH和rbcL基因的组合区别能力约为88%[21]。
总之,尽管在植物中没有发现类似于动物中的COI的单独的植物标记,但几个质体基因组的标记物已显示出作为与accD基因、ndhJ基因和rpoB基因相关的DNA条形码的优点,例如psbA-trnH、rbcL和matK、rpoC1[ 9,13,18,22,23 ](图1)。
这里有DNA条码的两组潜在用户:植物分类学家和其他领域的科学家[14,24]。此外,对非专业用户如海关官员、传统药物生产者和管理者、法医专家而言,DNA条形码将是一个有用且强大的工具。因此,一个快速、简便的DNA条形码识别系统,即使是不完美的,也很可能会受到欢迎。
在这项研究中,我们测试了七个潜在DNA区域(psbA-trnH、matK、rbcL、rpoC1、ycf5、ITS2和ITS)在8557种药用植物和密切相关的样品中作为DNA条形码的适合度,以上药用植物和样品属于5905种1010属219科7门(被子植物、裸子植物、蕨类植物、苔藓植物、藻类和真菌)。这些植物有着悠久的作为传统草药的使用历史,包括一些从中国和日本的药典,以及欧洲药典中获知的历史。这七个候选DNA条形码使用几个标准进行比较。四个位点(rbcL、matK、rpoC1和ycf5)被植物工作组 (www.kew.org/barcoding)推荐,至于其他三个位点,psbA-trnH和nrITS被Kress等推荐,ITS2被Chiou等人首先描述[25]。ITS2区被选择作为一个条形码候选是因为条形码ITS2序列具有一般系统发育标记的潜能,并被广泛用于系统发育属和种属的重建[ 26-30 ]。中草药的寻找和开发在全球范围内迅速增加,因此迫切需要实际并准确的身份验证资源[ 31-34 ]。我们的研究表明DNA条形码技术将成为鉴定潜力药用植物及其混淆品的一种标准。
成果
PCR扩增效率
4对ITS1引物的PCR扩增成功率在我们的试点研究中效果较差,所以ITS1没有被列入随后的实验。由Chiou等[25]设计的两对ITS2引物,一个专为这项研究设计而未能在裸子植物和蕨类植物的序列中扩增。成功率最高的引物对在每个条形码表S1中列出。我们通过实验研究获得了这些比率。例如,我们比较了psbA-trnH,ITS2 和ITS序列在400个样品中的PCR扩增效率,以上样品属于326种,98科,包括双子叶植物、单子叶植物、裸子植物和蕨类植物。psbA-trnH和ITS2序列的成功率分别为92.8%和93.8%,而其片段在实验中的扩增率只有42.3%(表1)。此外,我们还计算了所有样品的PCR扩增中样品总数的效率(表S2)。结果表明,rpoC1成功率最高,其次是psbA-trnH和ITS2。
用六个参数确定遗传差异
首先,三个参数被用来表征相互间特定的分歧[35,36]:(i)至少两个属中每个种内的所有个体的平均距离(K2P距离);(ii)平均theta;,theta;的意思是多于一个物种的每个属内的两两距离,这样可以消除不同数量的物种产生属间偏差;(iii)最小的相互间的距离,即至少两个物种的每个属间的最小距离。一个良好的条形码应该具有高的相互间的差异,以区分不同种类。同属的物种之间使用六个候选条形码比较种间遗传距离,根据所有三个参数之间的特定标准,ITS2区表现出较大的差异,其次是psbA-trnH序列(图2和表2),而 rpoC1最低。此外,Wilcoxon符号秩检验证实,ITS2和psbA-trnH序列提供了同属的物种之间最高的种间分歧,而最低的是rpoC1(表3)。
其次,三个附加参数被用来确定种内变异[ 18,35 ]:(i)平均比内差异(K2P距离),在收集样品时,每个物种收集多于一个个体;(ii)theta;,theta;表示在每个物种中的两两平均距离,至少采用两个代表;是在物种间取样时消除偏见与不平等;(iii)平均接合的深度,这是最大的在每个物种种内距离至少采用两个个体。在这里,采用三个参数确定种内变异时,rbcL基因表现出最低的水平,而ITS2仍表现出最高水平,其次是psbA-trnH序列(图2和表2)。针对种内变异,Wilcoxon讯号等级检验表明,rbcL、rpoC1和matK显示的种内变异最小,而ITS2最高(见表四)。
同样地,如上所述,使用六个参数和统计检验变异情况时,候选DNA条形码中ITS2和psbA-trnH被发现有高的种间差异和种内差异。这一分析表明,ITS2和psbA-trnH序列即是最符合我们要求的DNA条形码。他们进一步评估时,使用2个评估标准:DNA条形码技术差距和认证能力。
条形码差距评估
在理想情况下,一个DNA条形码的遗传变异应展示种间和种内样品的单独性,互不重叠分布。Meyer 和 Moritz 等的研究表明[35,37] ,当相近物种的数量增加时,遗传变异的重叠条形码差距也显著增加。我们的研究表明,在研究种内和种间差异分布时,psbA-trnH
和 ITS2呈现显著差异,但是当研究种内同种个体和种间差异时,所有细节之间的分歧使用matK基因,rbcL基因,ycf5和rpoC1检测,有显著不重叠间隙(图S1)。然而,计算同一个体的种内差异和同种物种的种间差异时,即物种的紧密程度增加时,条形码没有显示大的差异(图3和图2)。此外,Wilcoxon的两个示例测试表明,六个条形码(ITS2 psbA-trnH,matK,ycf5,rbcL和rpoC1)的种间差异均值明显高于相应的种内差异(图3)。因此,通过这个测试,psbA-trnH和ITS2能更好地显示种内和种间差异。
物种鉴定能力的评价条形码
使用DNA条形码技术的物种鉴定评估能力,具体有两种方法(BLAST1和最近遗传距离)被应用[ 12 ]。BLAST1方法确定一个样本的基础时,最合适的查询序列和用于匹配的E-值必须小于临界值。相比较而言,最近的遗传距离法确定一个样品时是基于该序列在数据库中的查询序列中有最小的遗传距离,该距离必须小于一个遗传阈值。我们首先检索所有的ITS2和psbA-trnH序列构建的参考序列库。然后,我们在数据库中搜索在这项研究中所产生的序列与成熟的样本分类标识。这些查询序列的物种身份是接下来用于决定是使用BLAST1还是最近遗传距离法的关键。
结果表明,使用BLAST1方法,在种和属的层次上,ITS2分别正确识别92.7%和99.8%的样品。当采用最近遗传距离法时,在种和属的层次上,ITS2分别正确识别90.3%和99.7%的样品(表3)。相反,在物种水平上使用BLAST1和最近遗传距离法时,ITS2和psbA-trnH的成功率非常低(分别为67.6%和72.8%)。然而,在鉴定到属的等级时,psbA-trnH序列有95%的成功率(表3)。我们的数据表明,当样品在进行植物类群广泛测试时,ITS2具有更高的成功率,在种属鉴定时,psbA-trnH的成功率更高。因此,ITS2区是一个功能强大的通用条形码,同时,它是一个很有前途的用于验证所有主要植物传统草药分类群的候选区。基因psbA-trnH间隔序列可作为一种补充条形码。
讨论
与其他研究相比,我们的研究针对ITS2区是鉴定药用植物最有前途的通用DNA条形码提出了一个强有力的案例,以此来作为评估的几个标准。首先,在160-320个碱基对中,ITS2区域较短。因此,ITS2序列是由我们组(表S1)选择的比较容易扩增的一对通用引物。其次,用六个参数确定遗传差异的统计试验证实,ITS2区具有较高的种间发散(图2,表2,S3)和分离作用。DNA条形码的差距和Wilcoxon两样本分析测试实验支持以下概念,即ITS2区的种间差异平均值明显高于种内差异的平均值(图3,S1-S3)。第三,根据本BLAST1方法,6685个样本(取自4800种753属193科)采用IT
剩余内容已隐藏,支付完成后下载完整资料

英语原文共 8 页,剩余内容已隐藏,支付完成后下载完整资料
资料编号:[286457],资料为PDF文档或Word文档,PDF文档可免费转换为Word




