高通量数据分析系统的设计与实现毕业论文
2020-04-04 10:53:00
摘 要
DNA测序技术是测定DNA序列的技术,目前用于测序的技术主要有一代测序、二代测序、三代测序技术等等。随着高通量测序技术的发展与深入,测序数据呈爆炸性增长。在分子生物学研究中,对DNA的测序分析是研究和改造基因的基础,如何快速高效地分析相关数据成为生物学家面临的一大难题,因此各种针对测序数据的生物分析流程也随之产生。生物分析流程的好坏直接决定了相关企业或科研院所的分析效率和能力。目前主流生物分析流程存在以下问题:1)分析过程非透明化,无法确保分析流程的完整性和准确性;2)许多并行化、分布式、云计算技术并没有应用到流程中,导致整体的分析效率不高;3)分析流程复杂性过高,目前很多分析都是在命令行下进行,对操作人员有更多的技术要求。
为了解决目前主流生物分析流程存在的问题,本课题要利用NextFlow框架实现一个自动化、并行化、有用户操作界面、可追踪分析过程的生物分析流程操作平台,致力于使用户掌握流程运行的状况,直接操作图形界面可以大大降低生物研究人员的操作难度,简化操作步骤,使生物流程分析更加高效。
本课题实现的系统主要包括数据管理模块、生物分析流程模块和报告模块。在数据管理模块实现了在可视化前端界面进行流程、配置文件及相关参数的选择,将选择的数据在后台拼装成命令以便在服务器执行;执行命令后进入生物分析流程模块,数据在nextflow自动并行工作流中经过层层处理,最后生成结果文件result.zip;结果文件在报告模块中首先被录入数据库,再从数据库中按需取出,经过数据可视化处理生成图表,最后生成动态报告。
关键字:自动化;并行化;数据分析;资源监控
Abstract
DNA sequencing technology is a technique for determining DNA sequences. The sequencing technologies include one-generation sequencing, second-generation sequencing and three-generation sequencing technologies currently. With the development and deepening of high-throughput sequencing technology, the sequencing data showed explosive growth. In the study of molecular biology, DNA sequencing analysis is the basis for the research and modification of genes. How to quickly and efficiently analyze related data has become a major problem fir biologist. Therefore, various biological analysis procedures for sequencing data will appear. The quality of the biological analysis process directly determines the analysis efficiency and ability of the relevant company or research institute. Popular bioanalytical processes have the following problems: 1) The analysis process is non-transparent and cannot ensure the integrity and accuracy of the analysis process; 2) Many parallel, distributed and cloud computing technologies are not applied to the process, resulting in the analysis efficiency is not high. 3) The complexity of the analysis process is too high. At present, many analyses are performed under the command line, and there are more technical requirements for the operators.
In order to solve these problems existing in the current bioanalysis processes, Nextflow framework is used to implement an automated, parallel, biometric analysis workflow operation platform with user interface and traceability analysis process. It is dedicated to enabling users to grasp the status of process operations and directly operate. The graphical interface can greatly reduce the operational difficulty of biological researchers, simplify the operation steps, and make the biological process analysis more efficient.
The system implemented in this project mainly includes data management module, biological analysis process module and report module. The data management module implements the selection of processes, configuration files, and related parameters on the visual front-end interface. The selected data is assembled into commands in the background for execution on the server; after the command is executed, it enters the bioanalysis process module, and the data is automatically parallelized in the nextflow. After processing layers in the stream, the resulting file result.zip is finally generated. The result file is first entered into the database in the report module, and then removed from the database as needed. After the data is visualized, a graph is generated, and finally a dynamic report is generated.
Key words: automated; parallel; data analysis; resource monitoring
目 录
摘要 I
Abstract II
第1章 绪论 1
1.1 研究背景和意义 1
1.2 研究现状 1
1.3 研究内容 2
1.4 论文的组织结构 3
第2章 Nextflow框架及可视化工具介绍 4
2.1 Nextflow框架 4
2.1.1 自动化、并行化 4
2.1.2 流程控制追踪 6
2.1.3 资源监控 8
2.1.4 缓存机制 10
2.2 可视化工具 11
2.2.1 Highcharts 11
2.2.2 D3.js 11
第3章 系统分析与设计 13
3.1 数据管理模块 13
3.2 生物分析流程模块 14
3.3 报告模块 15
第4章 数据管理模块 16
4.1 数据选择 16
4.2 后端处理 19
第5章 生物分析流程模块 22
5.1 编写nf脚本 22
5.2 编写运行配置脚本 25
5.3 执行流程 26
第6章 报告模块 30
6.1 数据录入 30
6.2 绘制图表 31
第7章 总结与展望 34
7.1 总结 34
7.2 展望 34
参考文献 36
致谢 37
第1章 绪论
1.1 研究背景和意义
基因测序技术是测定基因序列的技术,目前用于测序的技术主要有一代测序、二代测序、三代测序技术等等。随着高通量测序技术[1]的发展与深入,测序数据呈爆炸性增长。在分子生物学研究中,对DNA的测序分析是研究和改造基因的基础,如何快速高效地分析相关数据成为生物学家面临的一大难题,因此各种针对测序数据的生物分析流程[2]也随之产生。生物分析流程的好坏直接决定了相关企业或科研院所的分析效率和能力。目前主流生物分析流程存在以下问题:分析过程非透明化,无法确保分析流程的完整性和准确性;许多并行化、分布式、云计算技术[3]并没有应用到流程中,导致整体的分析效率不高;分析流程复杂性过高,目前很多分析都在命令行下进行,对操作人员有更多的技术要求[4];分析平台大部分功能需要收费,不能满足科研人员进行大量科学实验的使用要求。
高通量数据分析系统[5]可以使分析过程透明化,研究人员可以掌控流程中各个子流程的运行状况,通过Nextflow框架[6]输出的流程运行和跟踪报告,保证流程输出的最后结果的正确性;系统由于应用了并行化和分布式技术[7],大大提高了分析的效率,从而能够处理大规模的数据,得到更多的实验结果和更准确的规律;系统具有可视化操作界面,且可以监控流程运行情况和资源占用情况,使得研究人员可以更加方便快速地进行实验,得到数据分析结果,完成科研工作。
1.2 研究现状
当前国内外有不少生物分析平台,比如国内有华大基因的BGI Online、百迈克的BMKCloud、上海尔云信息科技公司的“云生信”平台,国外有Galaxy[8]、DNAnexux、BaseSpace等分析平台。
BGI Online,是华大基因与国内知名云服务商阿里云以及国际芯片制造商英特尔联手合作开发的一款基于“云”的生物信息数据云平台,它主要提供数据存储、数据传输、弹性计算资源以及生物信息分析方法的开发和共享。但是BGI Online是收费的,且价格比较高昂,科研工作者使用它进行科学实验来说是不太合适的。
BMKCloud,是百迈克公司开发的一款集成生物信息软件、数据库的生物数据信息分析平台。该平台对于不懂编程的人来说比较友好,可以一键完成生物分析,但是该平台同样是收费的。
Galaxy,是一个开放的生信分析平台。该平台允许用户无需下载安装其他分析软件,就可以进行科学分析实验,而且具有数据上传检索处理、序列分析等等分析功能。但是该平台的计算速度比较慢,不适合具有大规模实验数据和有效率需求的用户进行数据分析。
DNAnexux,这家公司主要成熟的产品就是打造云端DNA数据库,为不同的实验室提供安全、规范的云平台[9]。该平台侧重的是下一代测序的生物信息分析,而且只有部分功能可以免费使用,大部分功能收费。
1.3 研究内容
需要完成的工作主要包括:利用Nextflow框架将已有的分析流程自动化、并行化;设计分布式生物分析流程;实现可视化界面,能够对流程进行控制和追踪,能够对计算资源占用进行监控。
1.利用Nextflow框架将已有的分析流程自动化、并行化。在生物分析流程子系统中,使用Nextflow框架编写流程来处理数据。根据Nextflow框架自身的特性,可以实现数据流的自动并行计算[10]。Nextflow会自动将输入中的多个文件放进所设计的流程中进行相同的处理,这就实现了分析流程并行化。比如流程nf文件中,param.input="data/*.fa",流程就会将data目录下的所有fa文件分别作为流程输入进行分析,最后进行整合。又由于Nextflow框架是使用流程模块process以及管道channel来设计流程,输入经过一个流处理最终输出结果,整个过程只需nextflow run一条语句就可开始并自动进行,实现了自动化。
2.设计分布式生物分析流程。Nextflow框架本身就支持分布式,在流程的配置文件nextflow.config文件中,可以编写profiles配置来设置不同的运行环境,包括标准、集群和云。执行nextflow run命令时可以添加参数来指定该进程在本地、某个集群或是云服务器上运行。
3.实现可视化操作界面。使用web前端和后端的技术搭建报告子系统[11]。在报告子系统中,生物分析流程子系统生成的结果文件result.zip会被拆分然后录入数据库。编写数据可视化模块,从数据库中取出数据,利用可视化工具Highcharts和D3.js将数据可视化[12],将数据转化成各类统计图,生成统计报告。
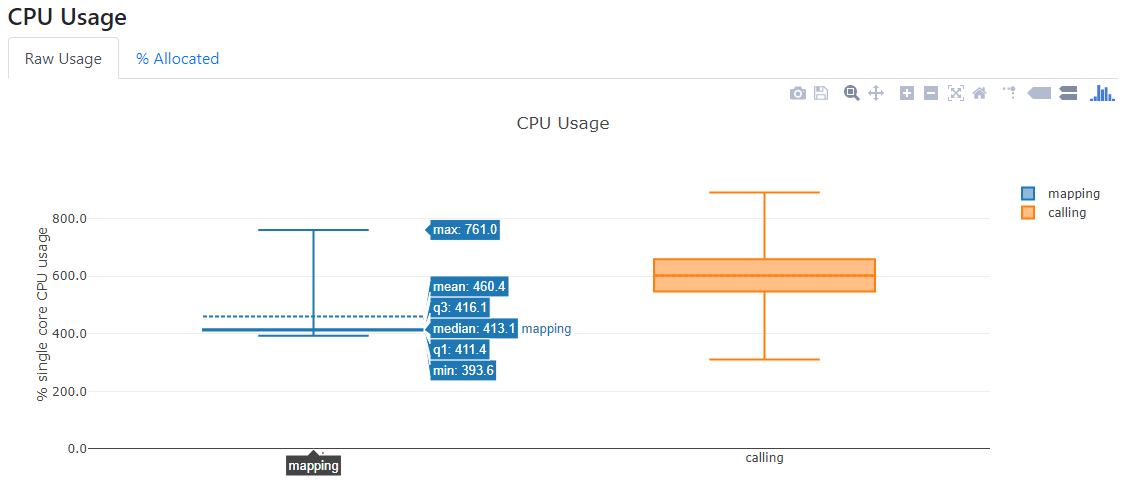
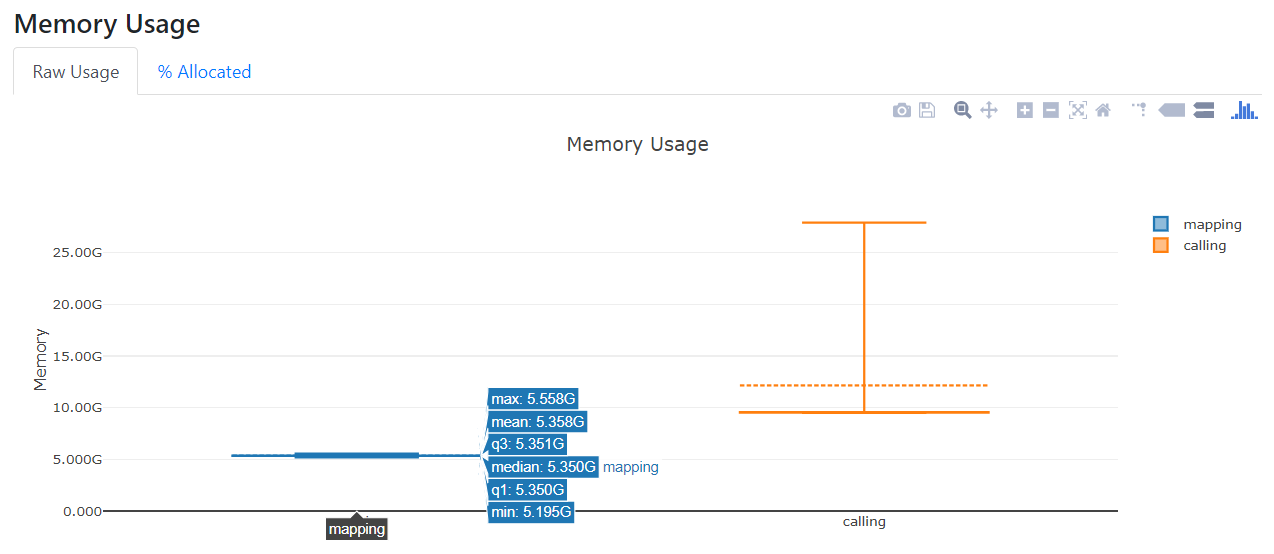
对流程进行控制追踪,对计算资源占用进行监控。根据Nextflow框架的特性,Nextflow在流程运行结束后,可以给nextflow run命令配置相应的参数来生成各种不同的报告。运行报告,可以展示每个工作流程的资源使用分布情况,通过配置-with-report参数可以生成此报告。跟踪报告,可以展示每个工作流程的一些有用信息,包括开始和完成时间、是否完成以及资源使用情况,通过配置-with-trace参数可以生成此报告。时间线报告,可以展示所有进程的时间轴,显示了各个进程之间的关系及依赖,通过配置-with-trace参数可以生成此报告。DAG可视化信息是nextflow流程通过DAG隐式建模,形成分析流程的流程图。根据这些报告,可以实现对分析过程的实时追踪和资源监控。
1.4 论文的组织结构
本论文主要分为7个章节:第一章为绪论,主要介绍研究背景及意义、研究现状、课题主要研究内容;第二章为技术介绍,包括Nextflow框架以及数据可视化工具;第三章为系统分析与设计;第四章为数据管理模块的实现,包括如何选择实验数据,如何将数据传输到生物分析流程;第五章为生物分析流程模块的实现,主要包括利用Nextflow框架设计流程;第六章为报告模块的实现,主要包括数据录入数据库,以及对数据进行数据可视化,形成动态报告;第七章是总结与展望,包括对系统实现进行总结,反思在完成过程中出现的问题以及对系统的下一步的改进。
第2章 Nextflow框架及可视化工具介绍
2.1 Nextflow框架
Nextflow是一个数据驱动[13]的计算流程框架,它使用软件容器实现了可扩展和可重复的科学工作流程[14]。用通俗一点的话说,Nextflow框架是用来构建工作流的,我们可以使用这个框架把一些脚本文件组织起来构成工作流,从而来处理和分析数据。
Nextflow框架有很多优点,比如说快速原型设计、可重复性[15]、跨平台、并行、具有连续的检查点、面向流的思想等等。
快速原型设计是指允许我们通过简化多个不同的任务来编写计算流程。可重复性是指Nextflow支持Docker和Singularity容器技术,而且集成了Github代码分享平台,我们可以进行版本管理,快速重现之前的配置。跨平台是指Nextflow提供了处于流程逻辑层和执行层之间的抽象层,所以无需改变就可以在多个平台上运行。并行是指Nextflow基于数据流编程模型,从而简化了编写复杂的分布式流程。Nextflow的并行化是由进程输入和输出声明隐式定义的。这样产生的应用程序在本质上是并行的,而且不需要适配特定的平台架构就可以扩展。具有连续的检查点是指流程运行中的所有中间结果都可以被自动追踪,这允许我们可以从上一次成功执行的步骤开始恢复执行,而不需要从头开始执行。面向流的思想是指Nextflow使用流畅的DSL扩展了Unix流程模型,使我们可以轻松地处理复杂的流交互。
2.1.1 自动化、并行化
Nextflow框架自动实现了自动化和并行化。
首先介绍进程和管道的概念。在Nextflow中,进程是处理用户脚本的基本处理单元。一个进程可能分别包含五个定义块:指令、输入、输出、条件和脚本。输入就是指传进该进程的数据,输出指的是进程要传出去的数据,脚本则定义了该处理单元所要执行的命令,对输入数据进行操作。由于Nextflow中各个进程彼此隔离,所以进程是通过管道进行通信的。管道是连接两个进程的非阻塞单项FIFO队列,管道发送消息通过异步的方式,无需等待接收消息的进程;接收消息时是阻塞操作,在消息到达之前都可以阻塞接收消息的进程。
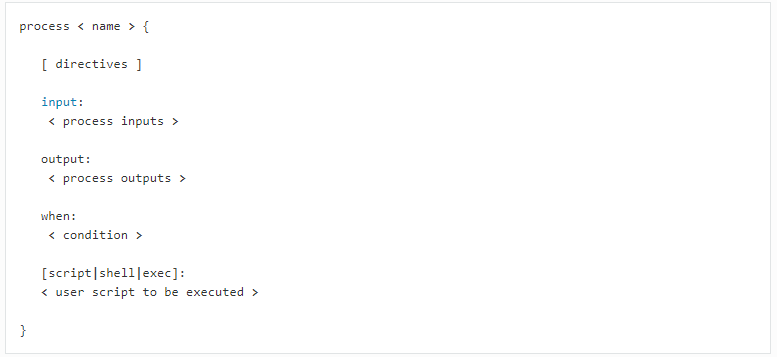
图2.1 process参数图
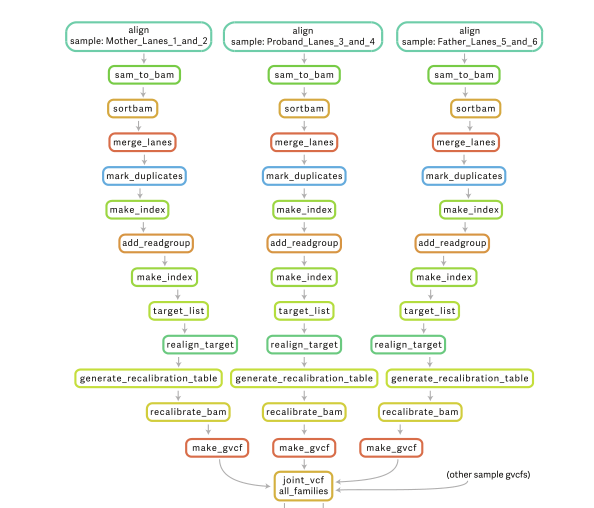
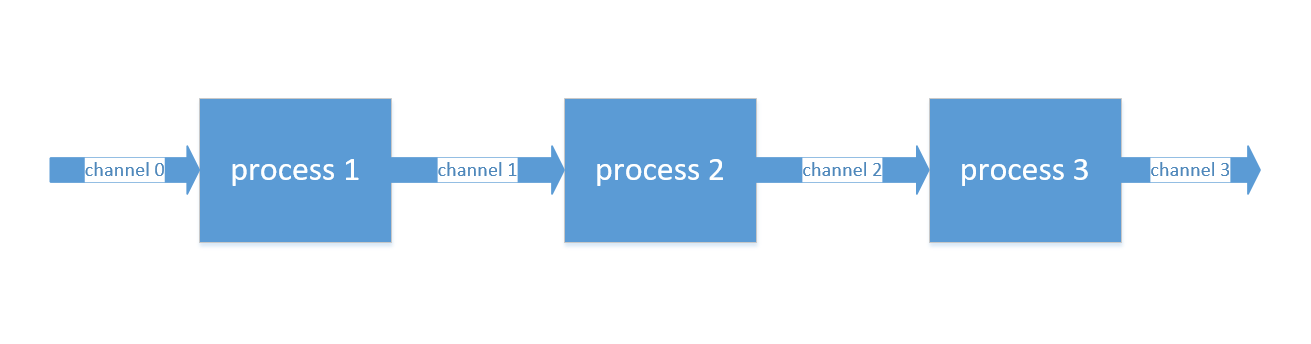
了解了进程和管道,就可以开始构建流程了。如图所示,是一个简单的流程。它是由三个进程和三个管道所组成。需要分析的数据通过第一个管道channel 0进入流程,channel 0会将数据传递进入第一个进程process 1,该进程的脚本中所包含的命令会处理数据,处理完毕后会将结果输出到管道channel 1;channel 1将结果传递进入第二个进程process 2,作为process 2的输入。接着就重复着类似的过程,直到最后一个进程process 3处理完数据,将结果输出到最后一个管道channel 3,整个流程就运行结束了。
以上是毕业论文大纲或资料介绍,该课题完整毕业论文、开题报告、任务书、程序设计、图纸设计等资料请添加微信获取,微信号:bysjorg。
相关图片展示: